COGNANOで機械学習に関する研究や開発を担当している鶴田です。先日、『アルパカ免疫により得られた抗原抗体相互作用の大規模データセットに関するCOGNANO・Googleの共著論文が世界最高峰のAI国際会議「NeurIPS 2023」に採択』というプレスリリースを公開しました。本論文では、アルパカという動物から創薬のためのデータを高品質かつ大量に創出するという非常にユニークかつ将来性のある方法論を提案しました。これは、COGNANOが目指す創薬の世界を実現するための出発点ともいえる研究であり、現在も論文中で示したコンセプトや技術に基づき、日々研究開発に取り組んでいます。今回は、我々のNeurIPS採択論文を機械学習(以下、ML)の観点から重要なポイントに絞って解説したいと思います。
Title | AVIDa-hIL6: A Large-Scale VHH Dataset Produced from an Immunized Alpaca for Predicting Antigen-Antibody Interactions |
Authors | Hirofumi Tsuruta, Hiroyuki Yamazaki, Ryota Maeda, Ryotaro Tamura, Jennifer N. Wei, Zelda Mariet, Poomarin Phloyphisut, Hidetoshi Shimokawa, Joseph R. Ledsam, Lucy Colwell, Akihiro Imura |
URL |
抗体創薬とAI
まずは、簡単に研究の背景についてお話しします。
抗体とは、我々の生命や健康を守る上で重要な役割を担うタンパク質です。人間は、外部から体内に侵入してきたウイルスや細菌などの抗原から体を守るために免疫という防御システムを備えています。この防御システムでは、体内に侵入した抗原に対抗するために大量の抗体を作り出し、抗原に結合して排除したり、機能を阻害したりします。この抗体を利用して、病気の予防や治療をおこなう薬が抗体医薬であり、抗体は抗原に対する高い結合親和性と特異性を持つことから、昨今重要な創薬モダリティの一つとなっています。抗体医薬の開発においては、標的抗原と抗体の候補との間の特異的な相互作用(これを抗原抗体相互作用という)を同定することが重要です。しかし、このプロセスは、非常にコストと時間のかかるバイオ実験に強く依存しています。
近年、従来の抗体探索のプロセスを加速するために、計算機を用いたアプローチに対する関心が高まっています。特に、AIやMLを使って計算機上で抗原抗体相互作用を予測しようとする取り組みが注目されています。もし、計算機のみで抗原抗体相互作用が予測できると、特定の標的抗原に対する結合抗体を仮想的にスクリーニングできるため、抗体探索の大幅な効率化が期待できます。例えばHuangら [1]は、抗原と抗体のアミノ酸配列を入力として、そのペアの相互作用の有無を予測する深層学習モデルであるAbAgIntPreを提案しました。抗体も多くの抗原もその実体はタンパク質であり、タンパク質は1文字のアルファベットで表記される20種類のアミノ酸が鎖状に並んだアミノ酸配列、つまり文字列として表現できます。AbAgIntPreは、二つの既存の公開データセット [2,3]を学習データに用いていますが、既存の公開データセットには、データ数が少ないことや、相互作用をしないペアのデータが存在しないこと、抗原の正確なアミノ酸配列が公開されていないことなどのいくつかの課題があります。そのため、今後AIやMLを活用した抗体創薬のさらなる推進には、大規模かつ高品質なデータセットの作成が必要不可欠です。
アルパカ免疫を利用したデータの生成
我々は、アルパカの免疫システムを利用した大規模かつ高品質な抗原抗体相互作用のデータセットの作成方法を提案し、実際に作成したデータセットであるAVIDa-hIL6を以下のサイトで公開しました。

注:CC BY-NC 4.0ライセンスの下に公開しているため、商用目的では利用できません。
ここでは、以下の図に沿ってデータセットの作成方法の重要なポイントを簡単に説明します。
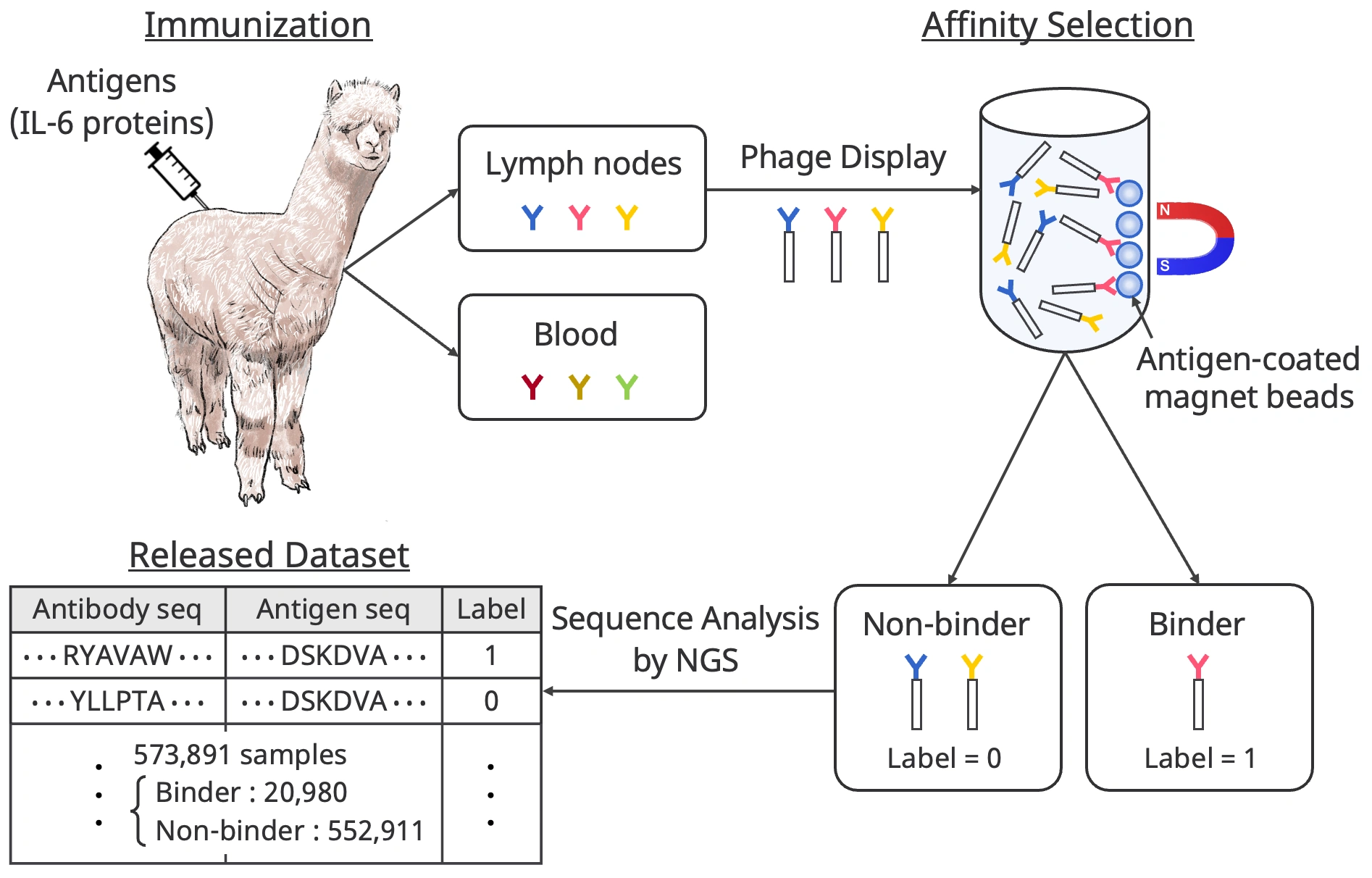
まず、特定の抗原をアルパカに注入します。今回の研究では、比較的単純な構造で多くの炎症性疾患や癌と関連しているIL-6タンパク質を抗原として用いました。抗原を注入すると、アルパカの体内では免疫反応が起き、抗原を撃退するための抗体が大量に作り出されます。
次に、アルパカからリンパ球や血液のサンプルを取り出します。このサンプルの中は、アルパカの免疫反応によって作り出したIL-6タンパク質に有効な結合抗体と、元々体内にあった有効でない抗体が混在している状態です。そこで、バイオパニングという親和性選択を行い、IL-6タンパク質に結合する抗体(Binder)と結合しない抗体(Non-binder)に振り分けます。これらの抗体のアミノ酸配列を次世代シーケンサ(NGS)という実験装置を用いて同定します。実際にBinderとNon-binderの振り分けは図で示しているほど簡単ではないため、我々が独自に設計した統計的な処理を用いています。
最終的なデータセットには、抗原(IL-6タンパク質)のアミノ酸配列、抗体のアミノ酸配列、抗原・抗体が結合するかしないかの2値ラベルの情報が収録されており、20,980件の結合ペアを含む573,891件のデータサンプルが含まれています。このデータ量は、我々の知る限り抗原抗体相互作用のデータセットとしては世界最大です。
なぜアルパカ?
ここまでの話で、そもそもなぜアルパカという動物を使っているのか、という疑問を持った方もいるのではないかと思います。我々の研究でアルパカを使っている主な理由は、最後のNGSでアミノ酸配列を同定するところにあります。結論を先に言うとアルパカは、効率的にアミノ酸配列を同定できる、つまりデータ化が容易な抗体を持っているからです。以下に、ヒトとアルパカが持つ抗体の構造の違いを簡単に示しました。
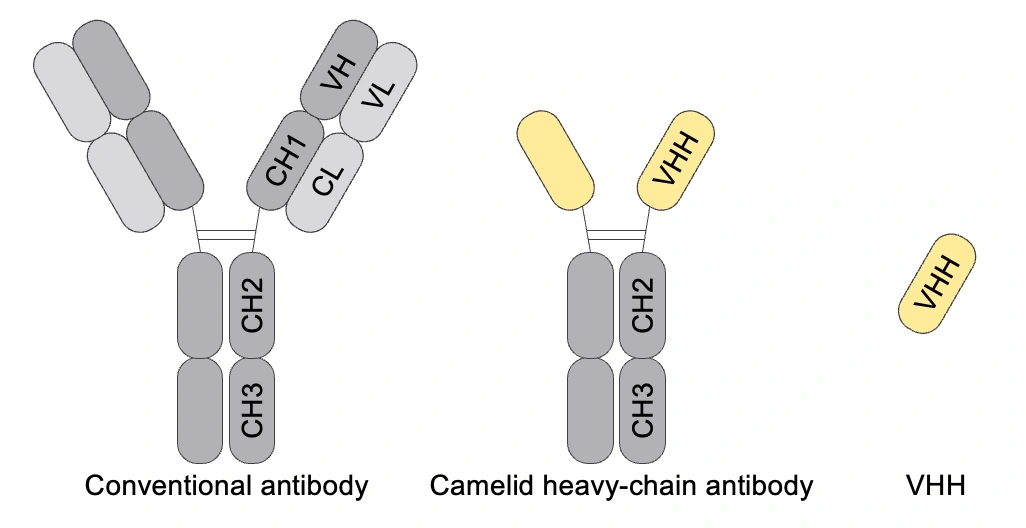
ヒトやマウスなどがもつ抗体(上図のConventional antibody)は2本の重鎖と2本の軽鎖からなります。一方、アルパカやリャマなどのラクダ科動物は、重鎖のみから構成される抗体をもつことが知られています(上図のCamelid heavy-chain antibody)。さらにその可変領域はVHH(またはNanobody)と呼ばれ、重鎖抗体の最小機能単位です。この単純な構造をもつVHHは、他の抗体に比べて、効率的にアミノ酸配列をデータ化できます。そのため、我々はアルパカをデータソースとして使用し、大量のVHHのアミノ酸配列データを作成しました。
抗原変異が抗体との結合に与える効果
ここまでで、アルパカのVHHを使うことで世界最大の抗原抗体相互作用のデータセットAVIDa-hIL6を作成したことを述べました。次に、AVIDa-hIL6の質の観点で重要な特徴を説明します。
新型コロナウイルスのパンデミックが示したように、ウイルスはヒトの免疫システムを回避するために変異を介して絶えず進化します。このような変異では、アミノ酸配列の一部が変化しており、わずかなアミノ酸配列の変化でも抗体との結合に大きな変化をもたらすことが知られています。そのため、抗原の変異が抗体との結合に与える効果を予測することは、治療用抗体の開発において重要です。
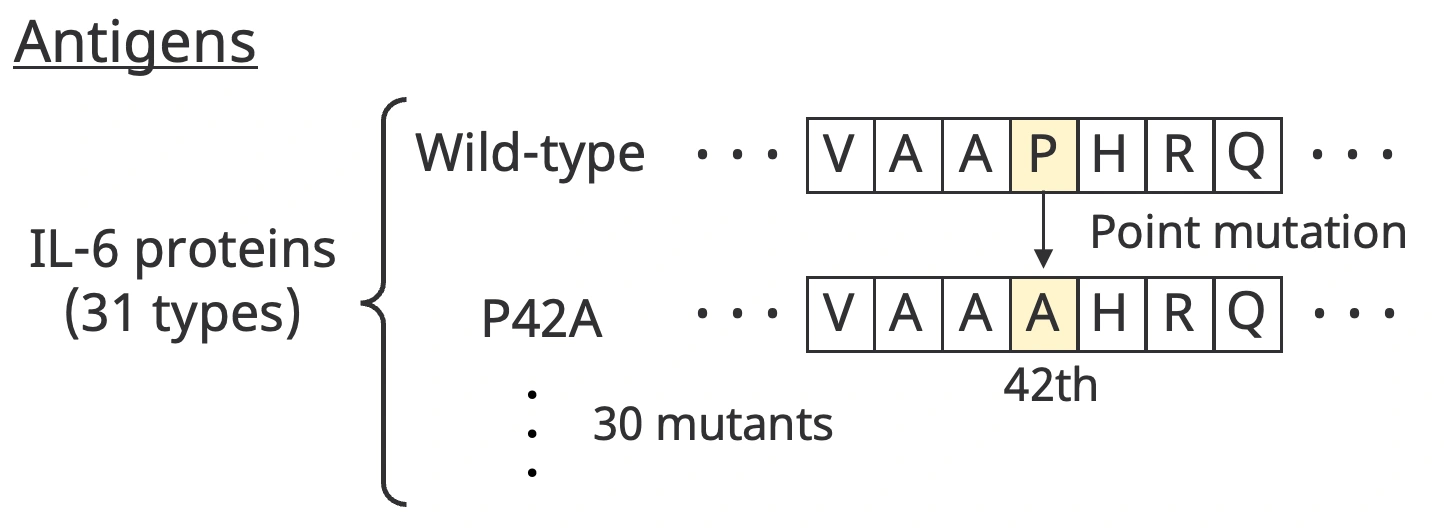
AVIDa-hIL6では、このような変異が抗体との結合に与える効果をMLモデルが学習できるようにデータセットを設計しました。そのために、抗原として野生型(wild-type)のIL-6タンパク質と、野生型の一箇所のアミノ酸を人工的に変異させた変異体を用いています。変異体は、野生型の30箇所の異なるアミノ酸を変異させた30種類の変異体を作成しました。例えば、以下の図のように「P42A」という変異体は、野生型の42番目のアミノ酸をP(プロリン)からA(アラニン)に置換したことを示します。
合計31種類のIL-6タンパク質をアルパカに免疫し、それぞれをターゲット分子として前述した親和性選択を行ったため、AVIDa-hIL6はアルパカが産出した多様な抗体が、それぞれのIL-6タンパク質の種類(以下、抗原タイプと呼ぶ)に結合するかしないかの情報を含んでいます。このようなデータセットの特徴を以下の二つの図を使って、もう少し詳しく見ていきます。
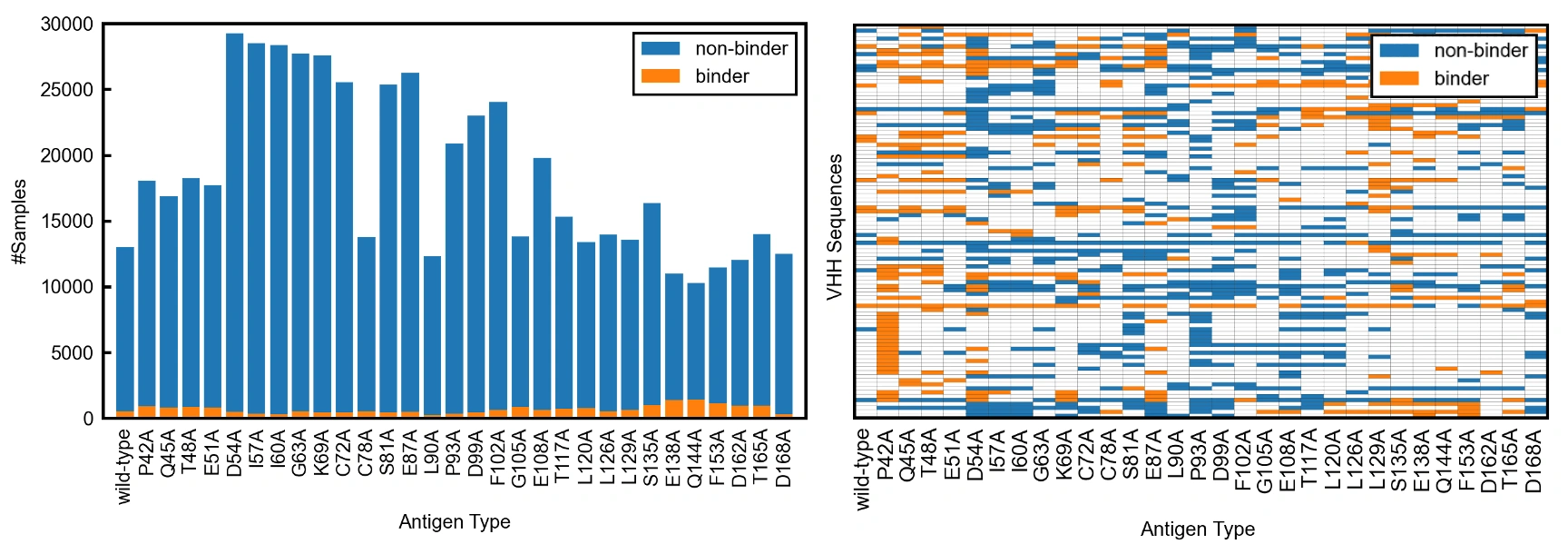
左図は、抗原タイプごとのデータサンプル数を示しています。抗原タイプごとに多少のサンプル数の偏りはありますが、最低でも250以上の結合抗体(binder)を含む10,000以上の抗体との結合情報のデータを持っています。
右図は、ランダムに抽出した100個の抗体がそれぞれの抗原タイプに結合するかしないかを可視化したものです。興味深いことに、AVIDa-hIL6には、抗原の1つのアミノ酸の変化により抗体との結合の有無が変化するような非常にセンシティブなデータが多く含まれていることが見て取れます。このような特徴を持つデータは、我々の知る限り他にありません。そして、この特徴は抗原変異が抗体結合にどのように影響するかを理解する上で重要な知見を提供しています。
データセットの利用例
論文中では、いくつかのMLモデルを用いて抗原抗体相互作用予測のベンチマーク結果を報告しています。
タスクの設計
AVIDa-hIL6の代表的な使い方は、抗原と抗体のアミノ酸配列を説明変数(入力)として、結合するかしないかの2値ラベルを目的変数(出力)とする2値分類問題を解くことです。一番シンプルなタスク設定は、全データから訓練データとテストデータにランダムに分割して、訓練データで学習したモデルを用いてテストデータに含まれる抗原・抗体ペアの結合の有無を予測することです。しかし、これでは抗原の変異体に対しての多様な抗体の結合データを持っているAVIDa-hIL6の特徴を活かすことができないため、抗原タイプに基づいてデータを分割することを考えます。実験シナリオとして、新型コロナウイルスのパンデミックのように、抗原の変異体が次々と出現することを想定します。そのような状況において、MLモデルがすでに観察された抗原の結合情報に従って、将来出現する未知の変異体に結合する抗体候補を予測できるかについて評価します。
このシナリオを実現するための実験手順を以下の図にまとめました。
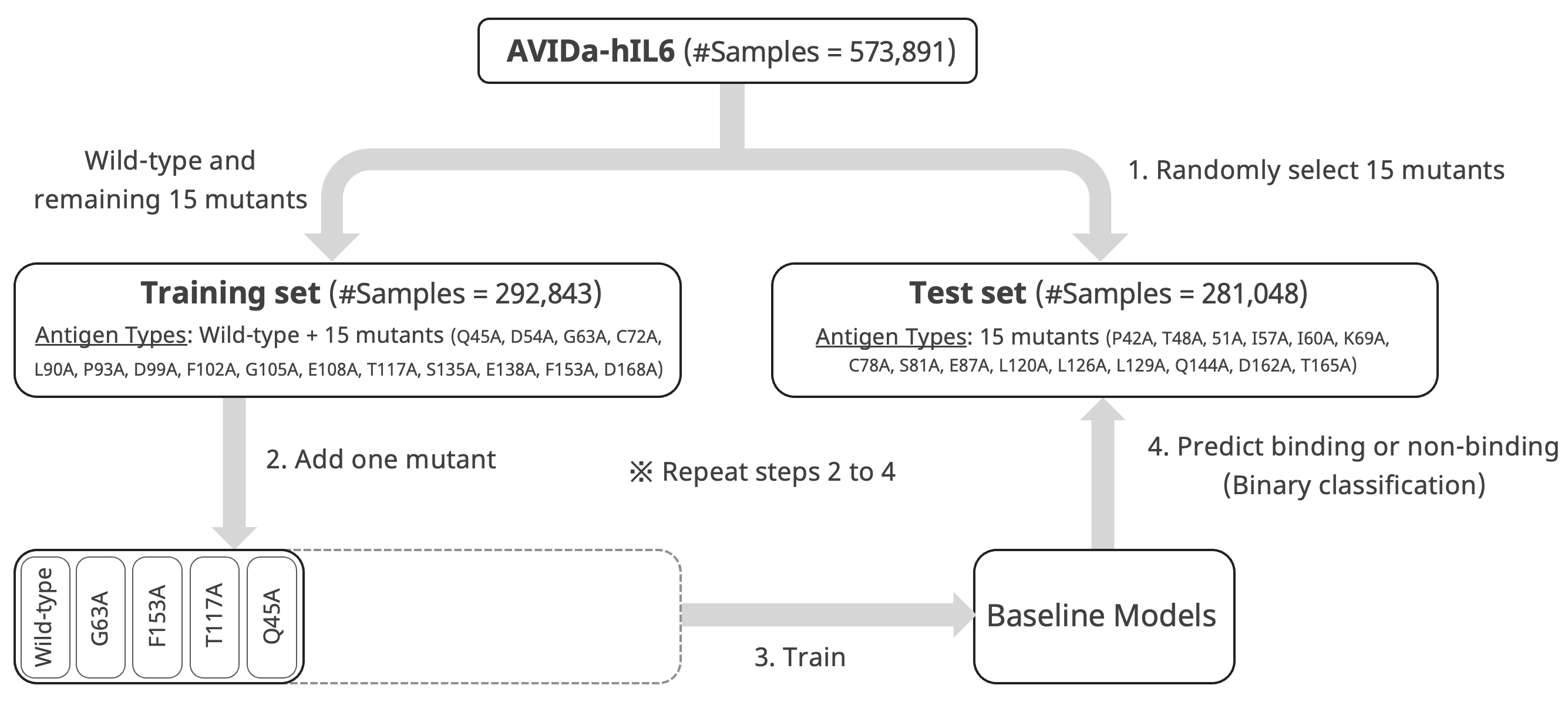
- まず、30種類の変異体のうち、ランダムに15種を選択し、それらの変異体を抗原とするサンプルをテストセットとして確保します。野生型と残りの15種の変異体に関するサンプルは訓練セットとします。
- 訓練セットから1つの抗原タイプをランダムに選択(一番最初は野生型で決め打ち)し、モデル学習用のデータセットに追加します。
- 学習用のデータセットを用いてベースラインモデルを学習します。
- 学習したモデルを用いて、テストセットに含まれる抗原・抗体ペアの結合・非結合を予測し、モデルの性能を評価します。
その後、2から4を繰り返し行うことで、MLモデルが学習に使える変異体に関する結合情報が増えた際に、未知の変異体(モデルが学習に使えないテストセットの変異体のこと)に対して有効な抗体を予測できるかを評価します。
ベースラインモデル
ベンチマークに用いるモデルとして以下の4つのモデルを採用しました。
- AbAgIntPre [1] AbAgIntPreは、2022年に提案された抗原抗体相互作用の予測のためのニューラルネットワークベースのモデルです。
- PIPR [4] PIPRは、2019年に提案されたタンパク質間相互作用の予測のためのニューラルネットワークベースのモデルです。
- Multi-Layer Perceptron (MLP) 1つの隠れ層を持つ非常に単純なニューラルネットワークベースのモデルです。
- Logistic Regression (LR) 2値分類タスクによく用いられる古典的なML手法です。
ベンチマーク結果
以下の図は、モデルの学習に用いた抗原タイプの数(横軸)とモデルの性能(縦軸)の関係を示しています。性能の指標として、左からPrecision、Recall、F1スコアを用いています。
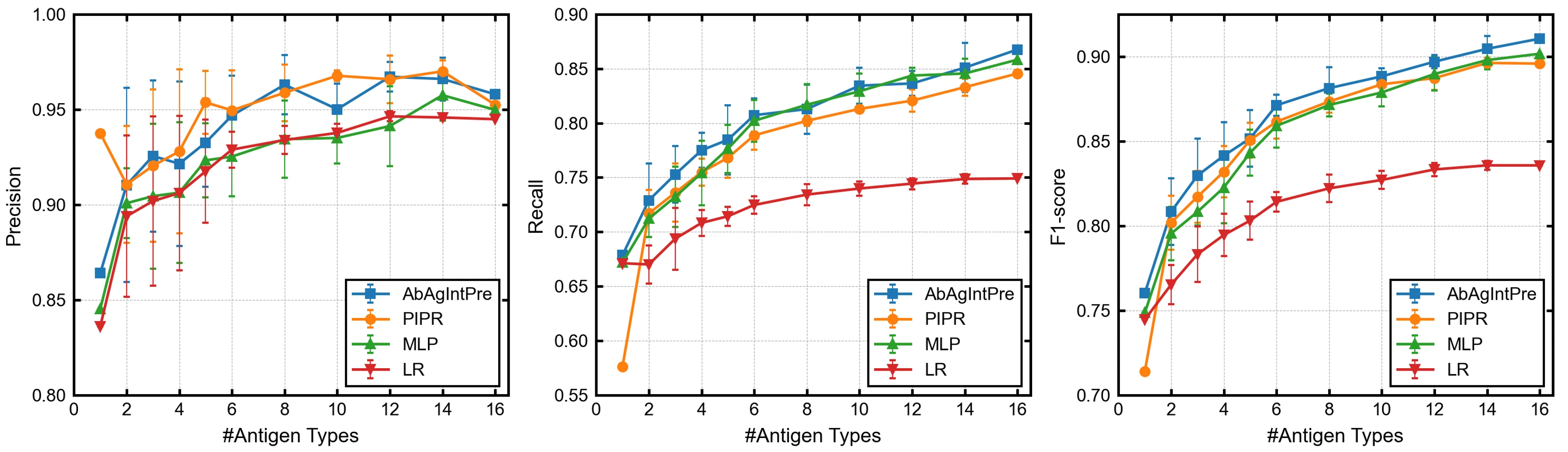
ここでは、Recall(モデルが結合抗体を予測できた精度)に着目して結果を見ていきます。まず学習に用いた抗原タイプ数が1の場合、つまり野生型のIL-6タンパク質のみを学習に用いた場合、AbAgIntPre、PIPR、MLP、LR のRecallはそれぞれ 67.9、57.6、67.2、67.1 %でした。この結果は、テストセットの変異体と結合する有効な抗体のうち30%以上を予測できていないことを意味します。次に、学習に使用した抗原タイプの数が増加するにつれて、Recallは増加しています。しかし、学習データに15種類の変異体を追加した後のRecallは、3種類のニューラルネットワークベースのモデルでも、85%程度でした。
AIやMLの創薬への応用では、できるだけ少ない抗原との結合情報から未知の抗原やその変異体に対して汎化性能を持つモデルを構築することが理想的です。そのことを踏まえると、まだまだRecallは高める余地がありそうです。これまでは抗原抗体相互作用の予測のための大規模なデータセットが存在しなかったため、抗原抗体相互作用の予測のためのモデル設計や開発はまだ発展途上だといえます。今回のベンチマーク結果は、抗原抗体相互作用の予測に特化したモデルアーキテクチャに関する更なる研究の必要性を提起しており、AVIDa-hIL6は、そのようなモデルの性能を評価するための有用なベンチマークとなります。
COGNANOの挑戦
本研究で提案したAVIDa-hIL6には、抗原の多様性が低いという大きな制約があります。具体的には、抗原として、IL-6タンパク質とその30種類の変異体しかありません。そのため、AVIDa-hIL6のみを使って学習したMLモデルが、IL-6タンパク質以外の抗原に対する抗体との結合を予測することは難しいでしょう。これでは、実用的なAI創薬の実現には程遠いです。しかし、本研究で提案したアルパカ免疫を用いたデータセットの作成方法は、アルパカに注入する標的抗原を変えることでIL-6タンパク質以外のあらゆる抗原に対して適用できます。実際に、同様のアプローチを用いて、SARS-CoV-2の変異体に関する抗原抗体相互作用の大規模なデータセットを作成し、有効な抗体の探索に成功しています [5]。
そのため、今後はより広範な抗原に対して同様のアプローチを適用し、より網羅的な抗原抗体相互作用のデータベースを構築していくことを目指しています。
最後に
今回公開したデータセットは、こちらのWebサイトから誰でも自由にダウンロードして使うことができます。ぜひ興味のある方は触ってみていただけると嬉しいです。COGNANOでは、AVIDa-hIL6以外にもまだ公開していないデータセットや、それらのデータセットを使った抗原抗体相互作用の予測に特化したTransformerベースのモデル設計・開発も進めています。ぜひCOGNANOの今後の取り組みにもご注目ください。最後までお読みいただきありがとうございました。
参考文献
[1] Huang, Y., Zhang, Z., Zhou, Y.: AbAgIntPre: A deep learning method for predicting antibody-antigen interactions based on sequence information. Frontiers in Immunology 13 (2022)
[2] Dunbar, J., Krawczyk, K., Leem, J., Baker, T., Fuchs, A., Georges, G., Shi, J., Deane, C.M.: SAbDab: the structural antibody database. Nucleic acids research 42(D1), D1140–D1146 (2014)
[3] Raybould, M.I., Kovaltsuk, A., Marks, C., Deane, C.M.: CoV-AbDab: the coronavirus antibody database. Bioinformatics 37(5), 734–735 (2021)
[4] Chen, M., Ju, C.J.T., Zhou, G., Chen, X., Zhang, T., Chang, K.W., Zaniolo, C., Wang, W.: Multifaceted protein–protein interaction prediction based on Siamese residual RCNN. Bioinformatics 35(14), i305–i314 (2019)
[5] Maeda, R., Fujita, J., Konishi, Y., Kazuma, Y., Yamazaki, H., Anzai, I., Watanabe, T., Yamaguchi, K., Kasai, K., Nagata, K., et al.: A panel of nanobodies recognizing conserved hidden clefts of all SARS-CoV-2 spike variants including Omicron. Communications Biology 5, 669 (2022)